- プロテアソーム活性化因子の機能解析
- Cullin型ユビキチンリガーゼの分子メカニズム
- 脱ユビキチン化酵素USP15によるRNAスプライシング制御と神経筋疾患との関連性
細胞内タンパク質分解について
細胞内タンパク質分解のシステムは、使い古されたタンパク質、すなわち寿命に達したタンパク質を取り除き、細胞内タンパク質の恒常性維持に貢献することが主な役割と考えられてきました。しかし最近の研究から、タンパク質分解は細胞内の恒常性維持のみならず、厳密な時空間的な制御を受けて、細胞周期、ストレス応答、発生、分化をはじめとする非常に広範な生命現象を積極的に制御することがわかってきました。これら多岐に渡る現象を制御するためには、特定のタンパク質を選択的に認識し、分解していくことが非常に重要になります。この選択的タンパク質分解に重要な役割を担っているのがユビキチンープロテアソームシステム(UPS)になります。
ユビキチン-プロテアソーム系のメカニズム
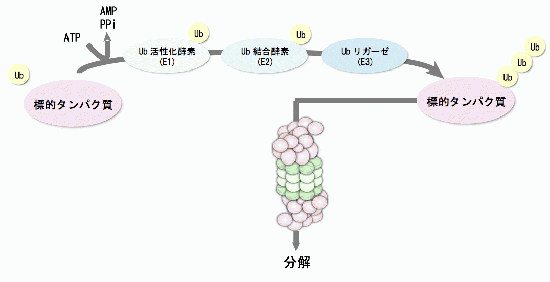
UPSは、E1、E2、E3の酵素群とプロテアソームからなる一連のカスケード反応からなりたっています。ユビキチンリガーゼ(E3)は、標的タンパク質にユビキチンを付加することでプロテアソーム依存的な選択的タンパク質分解を誘導します。生体内には非常に数多くのE3が存在し、それぞれのE3には特定の基質が存在することから、E3が標的の選択性を決定する上での鍵分子になると考えられています。
1. プロテアソーム活性化因子の機能解析
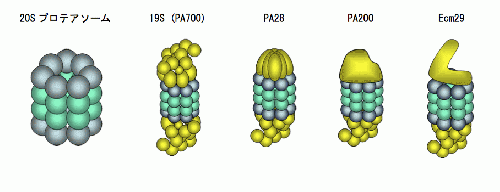
生体内における選択的なタンパク質分解は複合体型プロテアーゼであるプロテアソームにより行われます。20Sプロテアソームは、内部にプロテアーゼ活性部位を持つ樽状構造をとり、通常は入り口が塞がった不活性状態です。この活性はプロテアソーム活性化因子が20Sプロテアソームの樽状構造の片方または両端に結合することにより制御されています。活性化因子には19S, PA28αβ,PA28γ,PA200などがあります。またEcm29が結合タンパク質として知られています。しかし、それぞれの詳しい機能について未解明な部分が多くあります。私たちは、これらの活性化因子や結合タンパク質によるプロテアソームの制御機構、生体内での働きを明らかにするため、ノックアウトマウスを用いた解析を進めています。
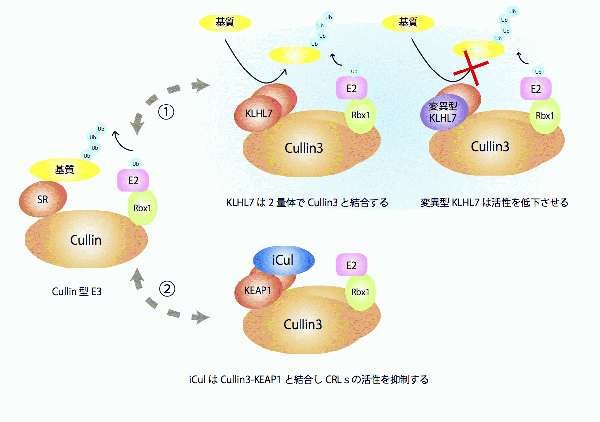
2. Cullin型ユビキチンリガーゼの分子メカニズム
CRLs (Cullin RING ligases) は足場タンパク質のCullin、触媒サブユニットのRbx1/2、基質認識サブユニットのSR (Substrate Receptor) からなる複合体です。本研究室ではiCulおよびKLHL7に着目し、CRLsの機能解析を行っています。
① 網膜色素変性症責任遺伝子 KLHL7
KLHL7はCullin3のSRです。私たちは、KLHL7の病原性変異がCullin3-KLHL7の活性を低下させることを示しました。しかし、KLHL7の機能や疾患メカニズムについては未解明な部分も多く、明らかにするために研究を進めています。
② 新奇ユビキチン化抑制因子 iCul
私たちはiCulがCRLsと複合体を形成し、Keap1をSRとするCullin3-Keap1の活性を抑制することを示しました。さらに、iCulが様々なCRLs関連因子と相互作用することも解ったので、新たな機構でCRLsの活性を制御する新たな因子としてiCulに注目しています。
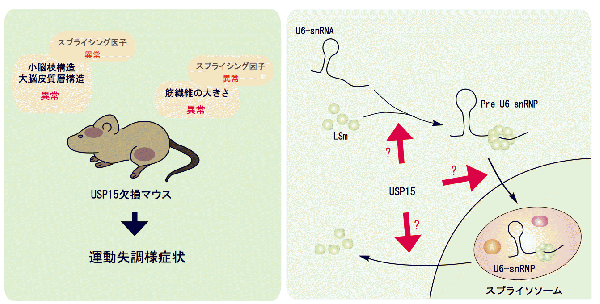
3. 脱ユビキチン化酵素USP15によるRNAスプライシング制御と神経筋疾患との関連性
これまでに私たちは、脱ユビキチン化酵素USP15のノックアウトマウスを作成し、機能解析を行ってきました。その結果、USP15のノックアウトマウスは振戦、歩行異常をはじめとする運動失調様の表現型を示すこと、USP15がRNAスプライシング制御に関わっていることを見出しました。最近、RNAスプライシング異常が、様々な神経変性疾患発症の原因となることが示唆されており、USP15のノックアウトマウスは、これら疾患に対する有益なモデルマウスになるのではないかと考え、研究を進めています。